3-mNGS有效鉴别胸腔积液:助力结核性胸膜炎及恶性肿瘤的诊断

导 读
近日,首都医科大学附属北京胸科医院病理科与迪飞医学合作,在Journal of Translational Medicine(IF=7.4)杂志发表一篇文章。该研究对138例胸腔积液患者进行mNGS和常规结核检测手段的性能比较,同时充分利用mNGS产生的人源序列和微生物序列,探究该技术在感染性疾病和鉴别恶性肿瘤中的价值。
研究背景
胸腔积液(pleural effusion, PE)是多种疾病常见的并发症,其病因诊断具有挑战性。常见病因包括结核分枝杆菌、肺炎旁胸腔积液和恶性肿瘤。
1.结核性PE抗酸染色阳性率非常低,PE的结核分枝杆菌培养阳性率为40%。分子诊断特异度高,但敏感性度低。
2.肺炎旁PE常继发于细菌性肺炎、肺脓肿或支气管扩张症合并感染。PE微生物学培养结果阳性是诊断肺炎旁PE的金标准,但培养周期长且阳性率约37.7%~53.5%。
3.PE细胞学检查诊断恶性肿瘤的敏感度为40.0%~87.0%,此外,PE也可作为液态活检样本进行宏基因组二代测序(metagenomic next-generation sequencing, mNGS) 检测,通过检测基因组不稳定性这一重要的恶性肿瘤遗传特征进行诊断。
因此,利用mNGS成为鉴别胸腔积液常见病因的有利工具。
研究亮点
1. 采用5种检测方法对结核性PE进行诊断,发现mNGS展示出优异的敏感性49%和特异性100%。
2. 利用人源序列进行拷贝数变异(copy number variant, CNV)分析诊断恶性胸腔积液(malignant PE, MPE),其灵敏度为54.1%,特异度为80.8%,准确度为71.7%,AUC为0.851。
研究路线
研究共纳入138例患者,其中包括结核性胸膜炎(tuberculous pleurisy , TBP)82例, MPE35例,非结核感染组21例。利用mNGS产生的微生物序列,评估mNGS对于结核的诊断价值。同时利用mNGS产生的人源序列,评估mNGS对于MPE的诊断价值(图1)。
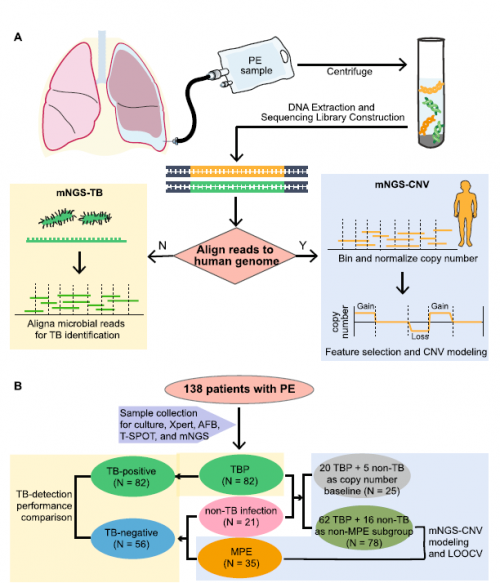
图1 mNGS对PE样本进行结核病检测和恶性预测的工作流程及入组队列展示
研究结果
不同结核诊断方法的性能比较,mNGS性能优异
本研究采用多种临床检测方法,包括培养、AFB实验、Xpert和T-SPOT,以及mNGS进行结核检测。
在所有临床检测中,T-SPOT检测显示出最高的阳性检出率,即61%(培养32%,Xpert 35%,AFB 12%),也略高于mNGS (49%,图2)。但T-SPOT法的假阳性率高达11%,远高于其他检测方法。
相比之下,mNGS展示出优异的敏感性(49%)和特异性(100%)。
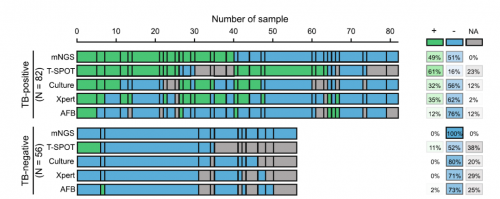
图2 mNGS与常规临床实验的结核检测结果
采用mNGS人源序列构建CNV模型识别MPE
为评估mNGS在鉴别恶性肿瘤中的价值,本研究采用GLM方法构建一个mNGS-CNV模型,以识别MPE。
随机选择25例非恶性胸腔积液(non-malignant PE , non-MPE)作为基线,对剩余113例(35例MPE和78例non-MPE)患者的染色体拷贝数进行分析并绘制拷贝数变异图谱,利用构建的mNGS-CNV模型来鉴别MPE和non-MPE(图3A)。与临床病理诊断相比,mNGS-CNV模型的灵敏度为51.4%,特异度为80.8%,准确度为71.7%(图3B,C)。
随后,通过LOOCV评估mNGS-CNV流程模型的预测效能, ROC曲线的AUC值为0.851(AUC值越接近1,表示模型性能越好;反之,AUC值越接近0,表示模型性能越差,图3D),显示出该预测模型的优异性能。

图3 mNGS-CNV分析的诊断性能。A. 具有代表性的MPE患者染色体拷贝数变异图;B. mNGS-CNV建模结果与临床病理诊断的比较;C. 基于B图的mNGS-CNV预测的敏感性、特异性和准确性;D. mNGS-CNV LOOCV结果的性能。
总 结
本研究充分利用了从mNGS中产生的微生物序列和人源序列,分析结果显示出mNG对TBP和MPE的良好鉴别能力,为mNGS同时进行病原体检测和肿瘤诊断提供可能,但未来仍需前瞻性临床研究和大样本验证。
该研究使用了迪飞医学mNGS检测技术。
参考文献:
Xu, F., Wang, Q., Zhang, N. et al. Simultaneous diagnosis of tuberculous pleurisy and malignant pleural effusion using metagenomic next-generation sequencing (mNGS). J Transl Med 21, 680 (2023). https://doi.org/10.1186/s12967-023-04492-x
